6.1.2.1 转录组测序样品制备方法
总RNA的提取与纯化方法同4.1.4.1。提取样品总RNA后,用带有Oligo(d T)的磁珠富集m RNA(Qiagen)。加入fragmentation buffer将m RNA打断成短片段,以m RNA为模板,用六碱基随机引物(random hexamers)合成第一条c DNA链,然后加入缓冲液、d NTPs、RNase H和DNA polymerase I合成第二条c DNA链,再经过QiaQuick PCR试剂盒纯化并加EB缓冲液洗脱之后做末端修复、加A并连接测序接头,然后用琼脂糖凝胶电泳进行片段大小选择,最后进行PCR扩增,建好的测序文库用Illumina HiSeq TM 2000进行PE90测序,测序数据量为5 Gb,测序流程图如图6-1所示。

图6-1 转录组测序实验流程图
6.1.2.2 转录组测序结果分析策略
转录组测序结果的分析流程图如图6-2所示,具体步骤如下:
1)数据产量统计
测序得到的原始序列数据称之为raw data或raw reads,在raw reads中含有带接头的,重复的,测序质量很低的reads。通过筛选过滤去除这些无效数据得到clean reads。
2)数据过滤处理的步骤
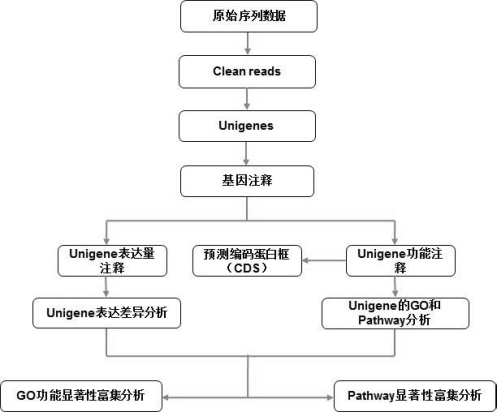
图6-2 转录组测序信息分析流程图
(1)去除含adaptor的reads;
(2)去除N的比例大于5%的reads;
(3)去除低质量reads(质量值Q≤10的碱基数占整个read的20%以上);
(4)获得Clean reads。
3)Clean reads序列拼接(https://www.xing528.com)
使用短reads组装软件Trinity[36]做转录组序列从头组装,Trinity首先将具有一定长度overlap的reads连成更长的片段,这些通过reads overlap关系得到的组装片段我们称之为Contig。然后将reads比对回Contig,通过paired-end reads能确定来自同一转录本的不同Contig以及这些Contig之间的距离,Trinity将这些Contig连在一起,得到两端不能再延长的序列,我们称之为Unigene。组装得到的Unigene,首先使用Tgicl将其去冗余和进一步拼接,然后再对这些序列进行同源转录本聚类,得到最终的Unigene(见图6-3)。
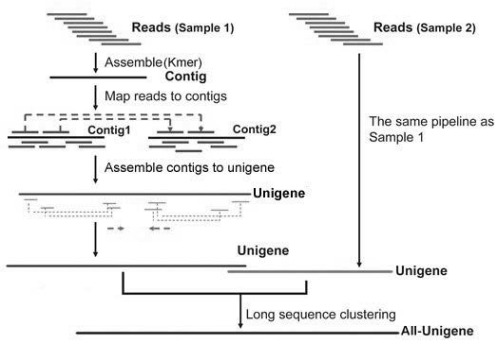
图6-3 序列组装步骤图
4)基因功能分析
(1)Unigene功能注释。
将Unigene序列与蛋白数据库Nr、Swiss-Prot、KEGG和COG做Blastx比对(e-value<0.000 01),得到跟给定Unigene具有最高序列相似性的蛋白,从而得到该Unigene的蛋白功能注释信息(功能注释、COG分类、KEGG Pathway注释),取比对结果最好的蛋白确定Unigene的序列方向。如果不同数据库之间的比对结果有矛盾,则按nr、Swiss-Prot、KEGG和COG的优先级确定Unigene的序列方向,跟以上四个库皆比不上的Unigene使用软件ESTScan[37]确定序列功能及方向。
(2)Unigene的GO分类。
根据nr注释信息能得到GO功能注释。Gene Ontology(简称GO)是一个国际标准化的基因功能分类体系,提供了一套动态更新的标准词汇表(controlled vocabulary)来全面描述生物体中基因和基因产物的属性。GO总共有三个ontology,分别描述基因的分子功能(molecular function)、细胞组分(cellular component)、参与的生物过程(biological process)。根据nr注释信息,使用Blast2GO软件[38]得到Unigene的GO注释信息。得到每个Unigene的GO注释后,应用WEGO软件[39]对所有Unigene做GO功能分类统计,从宏观上认识百子莲的基因功能分布特征。
(3)Unigene代谢通路分析。
KEGG是系统分析基因产物在细胞中的代谢途径以及这些基因产物的功能的数据库,利用KEGG可以进一步研究基因在生物学上的复杂行为。根据KEGG注释信息能进一步得到Unigene的Pathway注释。
(4)预测编码蛋白框(CDS)分析。
按nr、Swiss-Prot、KEGG和COG的优先级顺序将Unigene序列与以上蛋白库做blastx比对(evalue<0.000 01),如果某个Unigene序列比对上高优先级数据库中的蛋白,则不进入下一轮比对,否则自动跟下一个库做比对,如此循环直到跟所有蛋白库比对完。取blast比对结果中rank最高的蛋白确定该Unigene的编码区序列,然后根据标准密码子表将编码区序列翻译成氨基酸序列,从而得到该Unigene编码区的核酸序列(序列方向5'-3')和氨基酸序列。跟以上蛋白库皆比对不上的Unigene利用软件ESTScan[33]预测其编码区,得到其编码区的核酸序列(序列方向5'-3')和氨基酸序列。
(5)Unigene编码蛋白的Mapman生物学功能注释。
根据CDS分析获得的Unigene编码蛋白的氨基酸序列,提交到Mapman网站应用在线软件mercator(http://mapman.gabipd.org/web/guest/mercator)进行百子莲编码蛋白的生物学功能注释,并获得该物种的生物学过程的mapping信息。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。




