4.1.4.1 总RNA的提取与纯化
总RNA提取
(1)取2 g材料置于预冷的研钵中,液氮研磨至粉末状并转移至50m L离心管中(Axygen,RNase free),加入25 m L Trizol溶液(Invitrogen),震荡混匀后室温放置10 min。
(2)12 000 g 4℃离心5 min。
(3)小心吸取上清液,转入至新的离心管中;向上清液中加入1/5体积量的氯仿,盖紧离心管,剧烈震荡15 s,室温静置10 min,12 000 g 4℃离心15 min,小心吸取上清液至另一新的离心管中。
(4)向上清液中加入等体积的异丙醇,温和混匀,在室温下静置10 min,12 000 g 4℃离心10 min。
(5)小心弃去上清,缓慢地加入5 m L 75%的乙醇洗涤沉淀。
(6)12 000 g 4℃离心5 min后弃上清液;室温干燥沉淀2~5 min,加入适量的DEPC处理水溶解沉淀。
总RNA纯化
为防止基因组DNA影响后续试验结果,去除RNA中的基因组DNA污染,建立RNA纯化体系(见表4-1)。
表4-1 总RNA纯化体系

(续表)

(1)混匀后37℃水浴30 min。
(2)用50μL RNase-free H2 O定容至100μL后加入等体积的苯酚/氯仿/异戊醇(25∶24∶1)混匀。
(3)室温,13 500 rpm离心5 min,取上清液。
(4)加入等体积的氯仿/异戊醇(24∶1)混匀。
(5)室温,13 500 rpm离心5 min,取上清液。
(6)加入10μL 3 M醋酸钠,250μL冷乙醇,冰上放置10 min。
(7)4℃,13 500 rpm离心15 min,弃上清液。
(8)加入75%预冷的乙醇洗涤,13 500 rpm 4℃离心5 min,弃上清液。
(9)沉淀于室温干燥2 min,用适量的RNase-free H2 O溶解。
RNA完整性及质量检测
取5μL RNA样品用1%琼脂糖凝胶电泳分析,120 V电泳30 min,检测r RNA的带型,观察RNA的完整性。另取1μL RNA样品,用Nano Drop 1 000微量紫外分光光度计检测RNA浓度和质量。
4.1.4.2 mRNA 富集纯化
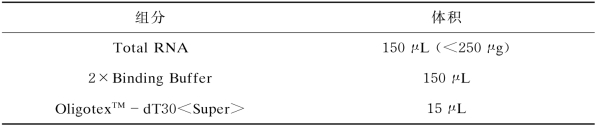
按照Oligotex TM-d T30<Super>m RNA Purification Kit(Takara)操作说明建立m RNA富集纯化体系(见表4-2)。
表4-2 mRNA富集纯化体系
(1)将上述配制的反应液充分混匀后,70℃加热3分钟(RNA变性);
(2)室温放置10 min(Oligotex TM-d T30和m RNA结合);
(3)15 000 rpm离心5 min;
(4)除去上清(尽量不要吸入Oligotex TM-d T30),加入350μL的Wash Buf fer,将Oligotex TM-d T30充分悬浮后,加入Spin Column Set的Column Cup中;
(5)15 000 rpm离心30 s;
(6)向Column Cup中加入350μL的Wash Buffer,使Oligotex TMd T30充分悬浮后,将Spin Column移至新的Spin Column用离心Tube上;
(7)15 000 rpm离心30 s;
(8)将Spin Column移至新的Spin Column用离心Tube上;
(9)向Column Cup中加入20~50μL 70℃预热的DEPC H 2 O,充分悬浮Oligotex TM-d T30。15 000 rpm离心30 s,回收m RNA;
(10)重复操作步骤(9)2~3次。
4.1.4.3 3’-RACE与5’-RACE cDNA文库构建
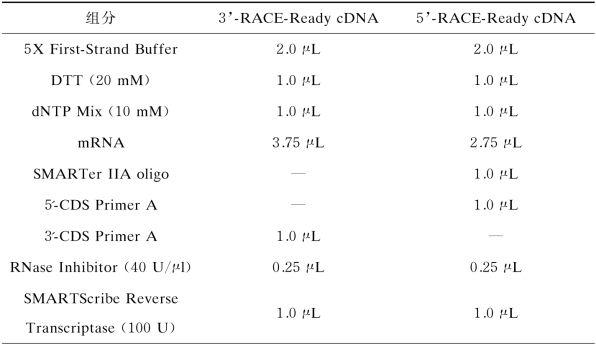
构建5’-RACE与3’-RACE c DNA文库按照SMARTer TM RACE c DNA Amplification Kit(Clontech)用户手册进行操作,文库构建体系如表4-3所示。
表4-3 3’-RACE与5’-RACE cDNA文库第一链合成体系
(续表)

(1)将上述配制的反应液充分混匀后,42℃孵育90 min;
(2)70℃处理10 min停止反应;
(3)用DEPC处理水将反应体系稀释至250μL,分装后放置于-80℃超低温冰箱中保存。
4.1.4.4 TIR1基因3’末端序列扩增
根据百子莲转录组测序获得的TIR 1基因序列(附录1)为参考设计特异性引物(TIR 1 sense,TIR 1 antisense),进行PCR扩增验证转录组测序获得的TIR 1基因拼接序列的准确性。根据验证过的TIR 1核心序列设计3’-RACE与5’-RACE的特异性引物。TIR 1核心序列引物及3’-RACE与5’-RACE的PCR引物如表4-4所示。
表4-4 3’-RACE与5’-RACE PCR引物列表
 (https://www.xing528.com)
(https://www.xing528.com)
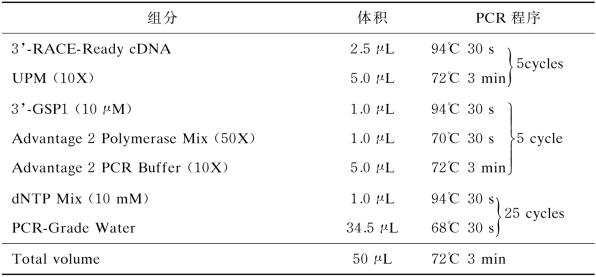
按照表4-5建立TIR 1基因3’-RACE PCR扩增体系。
表4-5 3’-RACE PCR体系
扩增产物检测:取5~10μL扩增产物用1%琼脂糖凝胶电泳检测(120 V,30 min),观察扩增效果。剩余产物用dd H 2 O稀释30~50倍于-20℃保存备用。
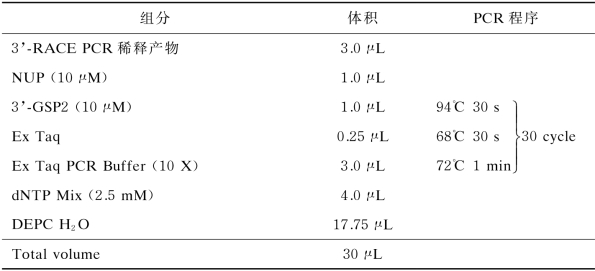
按照表4-6建立TIR 1基因3’-RACE巢式PCR扩增体系以获得3’-RACE特异性扩增片段。取20μL 3’-RACE巢式PCR扩增产物进行琼脂糖凝胶电泳检测,观察扩增效果。应用琼脂糖胶回收试剂盒对特异性扩增条带进行胶回收。
表4-6 3’-RACE巢式PCR体系
4.1.4.5 TIR 1基因5’末端序列扩增
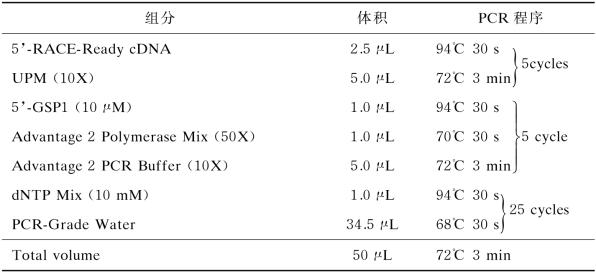
按照表4-7建立TIR 1基因5’-RACE PCR扩增体系。
表4-7 5’-RACE PCR体系
扩增产物检测:取5~10μL扩增产物用1%琼脂糖凝胶电泳检测(120 V,30 min),观察扩增效果。剩余产物用dd H 2 O稀释30~50倍于-20℃保存备用。
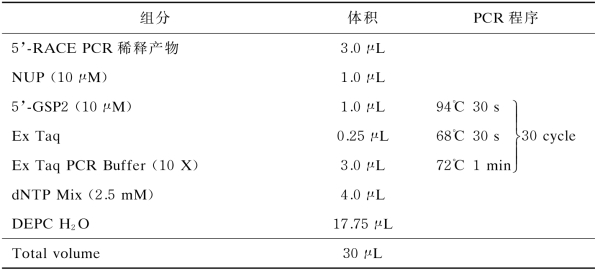
按照表4-8建立TIR 1基因5’-RACE巢式PCR扩增体系以获得5’-RACE特异性扩增片段。
表4-8 5’-RACE巢式PCR体系
取20μL 5’-RACE巢式PCR扩增产物进行1%琼脂糖凝胶电泳检测(120 V,30 min),观察扩增效果。应用琼脂糖胶回收试剂盒对特异性扩增条带进行胶回收。
4.1.4.6 3’-,5’-RACE目的片段的克隆与测序
1)DNA片段载体连接与转化
按照TaRa Ka公司的p MD 18-T vector使用说明,将回收的产物连接于载体上,反应体系如下(见表4-9)。
表4-9 载体连接体系

用枪头小心吹打混匀样品,16℃低温水浴中静置30 min。
2)感受态制备
(1)37℃震荡培养大肠杆菌DH5-α(200转/分)3 h;
(2)无菌条件下将1 m L菌液转移至1.5 m L Eppendorf管中,冰浴20 min;
(3)4℃10 000 r/min离心4 min,弃上清;
(4)加500μL遇冷的0.1 M CaCl2重悬菌体,置于冰上30 min;
(5)4℃10 000 r/min离心4 min,弃上清;
(6)每管加200μL 0.1 M CaCl2重悬,冰上放置备用。
3)连接产物转化DH5-α感受态细胞
(1)取200μL感受态细胞,在无菌条件下,加入连接反应液10μL;
(2)轻轻混匀内容物,冰浴放置30 min,其间每10 min轻轻混匀一下;
(3)将管在42℃水浴热激60~90s,不要摇动;快速将Eppendorf管移入冰浴中,冷却2~3 min;
(4)在超净台中每管加800μL已灭菌的LB液体培养基(见表4-10),置于37℃摇床振荡培养(180 r/min),60~90 min使细菌复苏,并且表达质粒编码的抗生素标记基因。
4)涂板
(1)复苏后的菌液4 000 g离心5 min,弃掉800μL上清液;
(2)用枪头小心吹打混匀剩余的200μL菌液(无菌条件下);
(3)在超净工作台上,将已经转化的感受态细胞全部转移到含氨苄青霉素(Amp+)的LB固体培养基平板上(见表4-11),用一个无菌的弯头玻璃棒轻轻将细胞均匀涂布,直至液体被完全吸收,用封口膜封口后,37℃倒置培养12~16 h。
5)蓝白斑筛选
待板上长出合适大小的蓝白斑后,用灭菌的枪头挑取5个阳性单克隆(白斑),并打入已灭菌的1.5 m L离心管中,管中预置1.0 m L含有氨苄青霉素(Amp+)的液体LB培养基(见表4-10),37℃,200 r/min摇菌,直至合适浓度。
表4-10 LB液体培养基配方∗
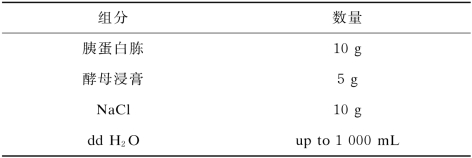
∗培养基经高温灭菌后,保存于4℃冰箱中备用。液体抗性培养基则在使用前加入氨苄青霉素(每1 L培养基中加入1 m L Amp+)。
表4-11 LB固体培养基配方∗

∗培养基经高温灭菌后,在超净工作台中冷却到60℃时每升培养基中加入1 mL Ampicillin(100 mg/m L)、1mLIPTG(24 mg/mL)、2 mLX-Ga l(20 mg/mL),混匀后倒入培养皿中,待培养皿中LB培养基彻底冷却凝固后封膜,保存于4℃冰箱中备用。
6)阳性菌落(白斑)PCR扩增鉴定
取2μL菌液做模板进行PCR扩增,PCR产物在1%琼脂糖凝胶上电泳分离;对于检测为阳性的菌落,将其相应菌液送到Invitrogen生物公司完成测序。
4.1.4.7 生物信息学分析
应用Clustal X(1.83)对测序获得的序列进行基因全长拼接。基因开放阅读框通过NCBI网站(http://www.ncbi.nlm.nih.gov/gorf/orfig.cgi)在线软件ORF Finder查询确定。蛋白质的基本性质分析及三级结构预测通过SOPMA与I-TASSER在线软件(http://zhanglab.ccmb.med.umich.edu/I-TASSER/)进行分析。蛋白3D结构应用Py Mol软件进行绘制。蛋白的疏水性由ProtParam软件分析(http://web.expasy.org/protparam/),蛋白见互作关系应用STRING软件分析(http://string-db.org/)。应用Blast X进行氨基酸序列比对,使用MEGA 5软件完成系统发生树的构建。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。




