1.全健康系统动力学模型技术应用
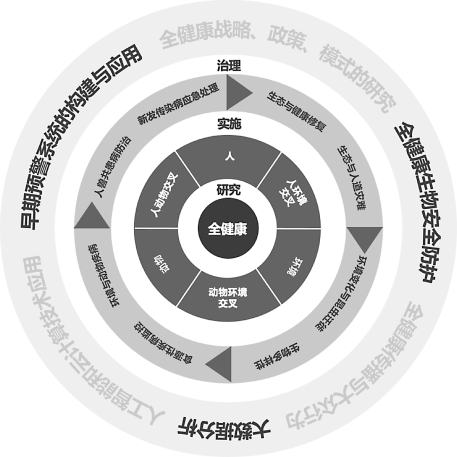
图1-2 全健康研究方向
数据的收集和整理是全健康理念的重要组成部分。通过系统动力学框架分析数据间的复杂关系并讨论其中的科学问题,是全健康相关课题中的重要组成部分。系统动力学是一门分析研究系统反馈互动作用过程的流行理论,也是一门认识系统问题和解决系统问题的综合学科。从系统论的观点看,系统动力学是结构方法、功能方法和历史方法的统一。它基于系统论,吸收了控制论、信息论的精髓,是一门综合自然科学和计算机模拟技术发展起来的学科。系统动力学最早由麻省理工的福瑞斯特(Forrester)教授于1956年创立,初期是用来解决生产管理及库存管理等问题的仿真模型,后来随着应用场景增大,可以用来分析各种传染病传播、生态系统变化、政策与实际效果的验证等情况[3],后由国内复旦大学王其藩教授等人将其引入国内[4]。近几年,系统动力学应用于传染病学和生态学的研究开始增多。其原因可能是数据平台的完善、监测技术的进步和病因网相关研究的深入,针对干预机制的研究也是其特点,经典案例有COVID-19和登革热[5-6]。在中国,系统动力学在全健康理念下的应用尚处于起步阶段,完整的系统动力学研究项目较少,需要科研人员一同建设和发展。目前,系统动力学模型主要靠VensimPLE或通过R语言建构。传染病类系统动力学模型的底层框架主要有两种。一类是易感人群、感染者及康复者(SIR)基础模型,包括易感人群、感染人群和暴露人群等参数;另一类是在SIR基础上扩展的模型,比如易感人群、传染者、感染者、康复者及死亡者(SEIRD)模型(图1-3),包括易感人群、暴露人群、感染人群、恢复人群和死亡人群等参数,该两种模型结构是传染病系统动力学模型的基础框架之一[7]。
2.环境DNA技术应用
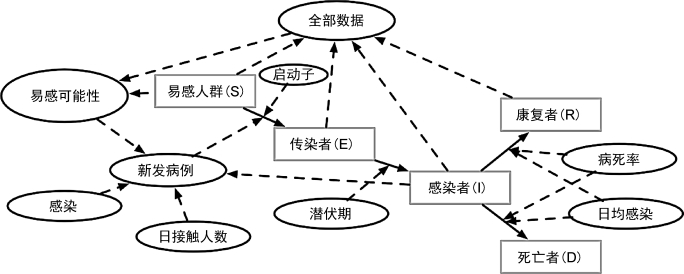
图1-3 SEIRD系统动力学基础模型框架(https://www.xing528.com)
1947年,美国生态学家道本迈尔(Daubenmire)将环境因子分为三大类:气候类、土壤类和生物类。其中非生物因子已经被研究得比较充分,有着较完善的监测网络,但生物类环境因子的检测还较为薄弱[8]。传统监测系统和系统动力学模型给全健康提供了基础框架,结合这些预测模型和客观事件,可以定位出传染病、外来入侵生物和耐药基因的低流行区,即“脆弱区域”,这也是监测框架中的关键区域。对脆弱区域进行的研究可以建立有效的早期预警模式。环境DNA条形码技术可以对真实世界进行针对性的实践,检验现有模型的可靠程度,是一个从宏观到微观的研究过程。比如,现有的传染病早期监测技术专注于在现有风险区域的易感生物和传播途径研究,缺乏针对真实世界“脆弱区域”的监测研究。以真实数据为基础的监控系统,能够得到更多的政策支持,真正实现早期监测预警框架的前移。环境DNA是从各种环境样板(如土壤、河水、雪和空气)中收集的DNA。结合现代测序技术,基于环境DNA的研究提供了一种非侵入性的方法,用于识别与DNA环境相关的物质或群落[9]。环境DNA最早应用于环境微生物学领域,被用于分离和纯化沉积物中微生物的DNA。自2003年环境DNA第一次被用于检测环境样本中的大型生物后,环境DNA的应用发展迅速,现已涉及食品微生物、人兽共患病监测和入侵生态学等多个领域[10]。
(1)在外来入侵生物研究中的应用:2012年,Takahara等率先采用环境DNA对日本淡水湖中的鲤鱼进行生物量分析[11]。2015年,Michelle等评估了环境DNA技术对水生外来入侵植物检测的可能性,结果表明环境DNA对水生植物监测可行[12]。2017年,Alba博士的研究表明环境DNA条形码技术可应用于外来入侵生物的鉴定和分类,可以提供足够的信息帮助当地进行保护规划和管理[13]。
(2)在生物监测中的应用:2019年,哥本哈根大学Mita教授的研究表明,环境DNA技术能够在水样中检测到钉螺和血吸虫,并且能够检测到血吸虫的不同生长阶段(毛蚴、尾蚴和虫卵)[14]。该实验结果表明,环境DNA检测的灵敏度和特异性均较高,适合真实世界的应用和实践(图1-4)。2020年,哥本哈根大学针对牡蛎相关寄生虫的实验也表明,可以从水环境DNA样本中检测到牡蛎包纳米虫的DNA[15]。Merou研究员关于牡蛎的论文对其检测灵敏度进行了更深入的研究,超过90%的寄生虫残留物在牡蛎离水2d后无法被检测出来[16]。
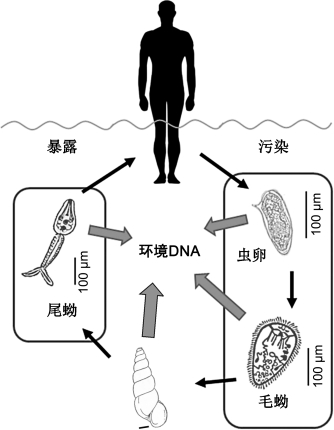
图1-4 血吸虫生活史示意图与环境DNA
(3)在耐药基因监测中的应用:细菌的耐药最早大多由医疗中的药物滥用导致,随着农业的集约化生产,兽医学也引入了抗生素,细菌对抗生素的耐受很快出现了。耐药基因存在水平基因转移,研究表明通过环境DNA也可以对抗生素耐药基因(antibioticresistantgene,ARG)进行检测和溯源[17]。这方面的研究集中于农业和污水处理,对ARG进行检测和溯源有助于维护粮食安全和食品安全,并且为可能存在的水平基因转移提供预警。
(4)环境DNA的测定:主要有聚合酶链反应(polymerasechainreaction,PCR)和DNA宏基因组两种方法。现有研究大多基于PCR进行特异性扩增,使用DNA宏基因组技术的较少,可能是基于敏感度和成本两方面的考虑。虽然,目前DNA宏基因组测序的单次检测成本较为昂贵,但随着技术进步,成本将有望变得可以接受。尽早建立操作标准和实验分析,进行可操性强的基础研究有助于扩大环境DNA在国内的影响力,推动行业发展。如果能够实现单次抽样检测多种类对象,将显著提升工作效率并降低检测成本。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。




