1.主要材料与设备
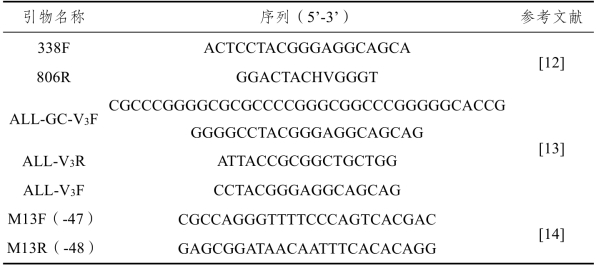
主要试剂:尿素、聚丙烯酰胺、N,N-亚甲基二丙烯酰胺、过硫酸铵、冰醋酸、甲醛、硝酸银、乙醇和十六烷基三甲基溴化铵购于国药集团化学试剂有限公司;QIAGEN DNeasy mericon Food Kit提取试剂盒,购于德国QIAGEN公司;10×PCR Buffer、dNTPs Mix、DNA聚合酶、pMD18-T vector和SolutionI,购于宝生物工程(大连)有限公司;Loading buffer、DL2000和DL15000 DNA Marker,购于宝日医生物技术(北京)有限公司;2×PCR mix,购于南京诺唯赞生物科技有限公司;Axygen清洁试剂盒,购于北京科博汇智生物科技发展有限公司;大肠杆菌Top10感受态细胞,本实验室制备。引物338F/806R(正向引物中加入7个核苷酸标签)、ALL-GC-V3F/ALL-V3R、ALL-V3F/ALL-V3R和M13F(-47)/M13R(-48)由武汉天一辉远生物科技有限公司合成。各引物序列如表1-5所示。
表1-5 引物名称和序列信息
HBM-400B拍击式无菌均质器:天津市恒奥科技发展有限公司;5810R台式高速冷冻离心机:德国Eppendorf公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司;DYY-12电泳仪:北京六一仪器厂;VeritiTM96孔梯度PCR扩增仪:美国AB公司;DCodeTM System:美国Bio-Rad公司;UVPCDS8000凝胶成像分析系统:美国BIO-RAD公司;MiSeq PE300高通量测序平台,美国Illumina公司;R920机架式服务器:美国DELL公司。
2.实验方法
(1)样品采集
从湖北省恩施市舞阳坝菜市场(109.47°N,30.3°E)采集5个腊鱼样品分别置于无菌采样袋中低温运回实验室。只有符合下列条件的样品才可纳入采集的范围:① 腊鱼由草鱼制作;② 腊鱼无肉眼可见杂质、寄生虫和虫卵;③ 腊鱼无病变、霉变和酸败等腐败变质现象;④ 腊鱼的制作地点需在恩施市范围内。
(2)样品前处理及宏基因组DNA提取(https://www.xing528.com)
取25 g切碎的腊鱼加入225 mL无菌生理盐水后置于拍击器中拍击3 min,取出置于冷冻离心机中300 r/min离心10 min保留上清液,上清液以10 000 r/min离心10 min以便收集菌体,然后使用商业试剂盒提取腊鱼样品微生物宏基因组DNA。
(3)基因组DNA检测
使用1%的琼脂糖凝胶电泳以DL15000Maker为参照查看是否扩增出清晰明亮大小合适的目的条带。然后以无菌超纯水为参照,取1 μL提取DNA置于微量紫外分光光度计检测口检测其OD260/280是否在1.8~2.0之间并确定各样品DNA浓度。用无菌超纯水将检测合格的样品宏基因组DNA浓度统一稀释至30 ng/μL备用。
(4)细菌DGGE指纹图谱分析
细菌PCR扩增时使用引物对为ALL-GC-V3F/ALL-V3R,扩增体系中各试剂添加量及循环条件参照文献15中的方法略做改动:细菌PCR扩增体系为50 μL,模板量为1 μL,退火温度(Tm)为55 °C。扩增结束后使用(2)中的方法检测扩增效果。在检测合格的体系中加入6 μL Loading buffer混匀置于-20 °C备用。DGGE凝胶中变性剂范围为35%~52%,待电泳槽中缓冲溶液的温度升至60 °C时在每个胶孔中加入10 μL混有Loading buffer的PCR产物,先120 V电泳76 min使样品穿过上层胶后80 V维持13 h然后采用银染法染色。取优势条带回溶溶液2 μL为模板进行PCR扩增,PCR扩增体系为25 μL:正反向引物ALL-V3F/ALL-V3R各0.5 μL,2×PCR mix 12.5 μL,剩余体积用无菌超纯水补齐。扩增循环条件及检测方法同上。参照杨春丽的方法将清洁后的扩增产物连接PMD18-T载体[16],并采用热激法[17]将连接产物导入大肠杆菌Top10中,挑选阳性克隆送去测序。
(5)细菌MiSeq高通量测序分析
首先对细菌16s rDNA进行PCR扩增,扩增方法参照文献18中所描述的方法进行操作。扩增合格的DNA使用干冰寄至上海美吉生物医药科技有限公司进行MiSeq高通量测序。参照蔡宏宇[19]的方法对返回的序列进行质量控制并根据核苷酸标签(barcode)区分序列来源。然后利用QIIME(v1.7.0)分析平台[20]对序列进行生物信息学分析:采用UCLUST法[21]划分分类操作单元(Operational Taxonomic Units,OTU),使用RDP(Ribosomal Database Project)[22]和Greengenes[23]数据库对OTU中各代表性序列进行同源性比对确定其微生物分类地位,使用超1(Chao1)指数和香浓(Shannon)指数分析腊鱼样品中细菌多样性。
(6)多元统计学分析
使用BioEdit 7.0.9和Mega7.0处理DGGE序列并绘制系统发育树,使用OriginPro 2017软件绘制其他图形。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。




