目前为止,绝大多数已发布的DNN的应用都涉及数据分类问题。这些应用绝大多数涉及图像和信号处理。本节讨论DNN模型在健康相关的领域上的应用。通过这个案例研究,可以放开思路,结合相关专业知识,将这个应用转化到科研领域中。
【例11.5】使用mbench包中的PimaIndiansDiabetes2数据构建DNN模型。
(1)数据集
数据集与糖尿病、消化和肾脏疾病研究有关,它包含768个观测值,每个观察值有9个变量,这些变量来源于21岁以上的印度传统Pima女性。表11.3是对数据集变量的描述。
<data("PimaIndiansDiabetes2",package="mlbench") #数据加载
>ncol(PimaIndiansDiabetes2) #列数
[1]9
>nrow(PimaIndiansDiabetes2) #行数
[1]768
表11.3 PimaIndiansDiabetes2数据框
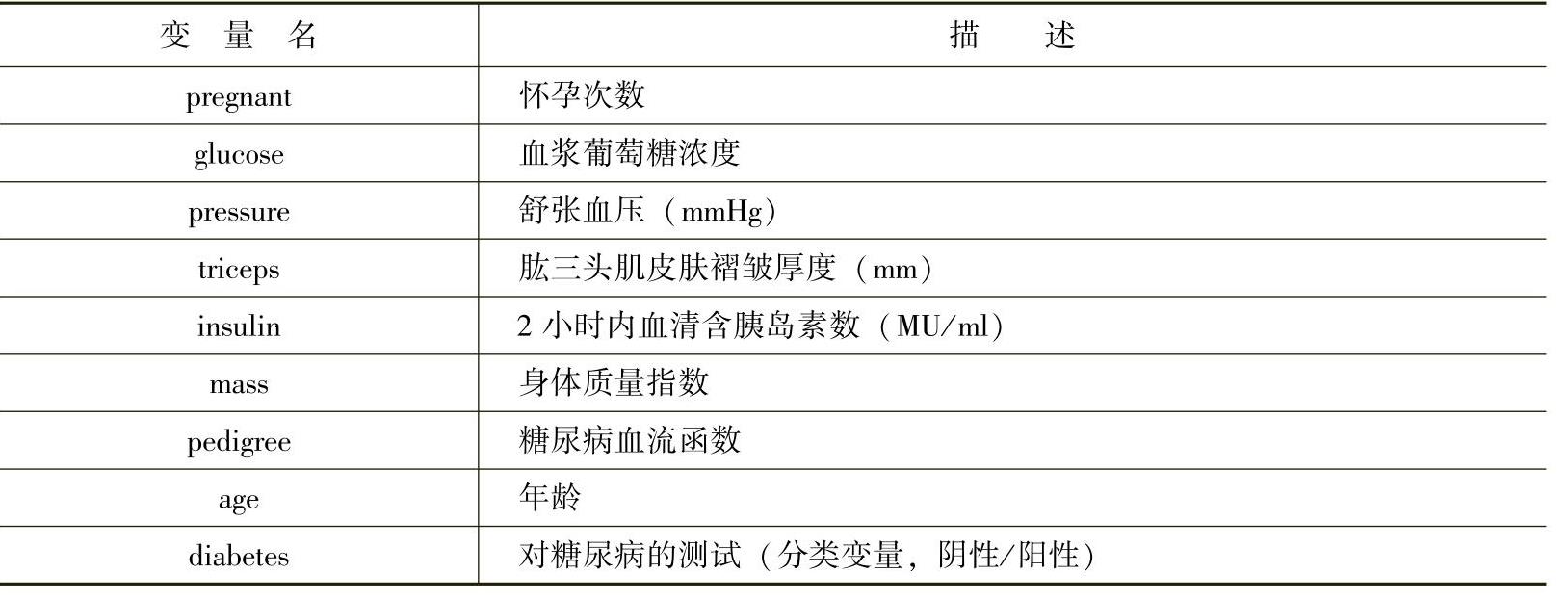
diabetes为响应变量。注意到,压力、肱三头肌、胰岛素和质量包含NA,这些是缺失值。收集数据时可能面临着丢失数据的问题,如人们不愿或忘记回答问题,因此,数据常常丢失或者无法被正确记录。这里数据似乎缺失很多,最好检查一下实际缺失的数据。
<apply(PimaIndiansDiabetes2,2,function(x)sum(is.na(x)))
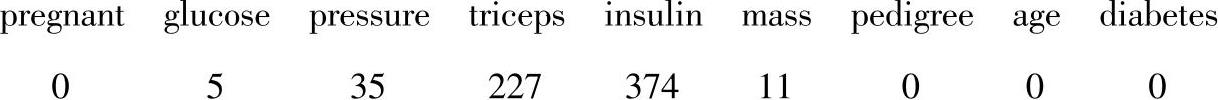
数据存在大量缺失值,特别是胰岛素和三头肌的属性。应该如何处理这个问题?最常用的也是最容易的方法是仅使用那些有完整个人信息的数据。用一个合理的值估算缺失的观测值也是一个替代方法。例如,可以用属性均值或中数替换NA。更复杂的方法是使用分布模型(例如最大似然和多重插补)。
由于胰岛素和肱三头肌缺失太多,从样本中删除这两个属性,并使用na.omit方法删除剩下的缺失值。被删除的数据存储在temp中。
<temp<-(PimaIndiansDiabetes2)
<temp$insulin<-NULL
<temp$triceps<-NULL
<temp<-na.omit(temp)
要做出有意义的分析,必须拥有足够的观测值。因此,考查一下数据:
>nrow(temp)
[1]724
>ncol(temp)
[1]7
数据包含了724个数据,响应变量存储在R对象y中,并从temp中删除它。注意,现在矩阵temp中只包含变量属性。使用缩放的方法将属性数据标准化;然后将y(作为因子)合并到temp中。
<y<-(temp$diabetes)
<temp$diabetes<-NULL
<temp<-scale(temp)
<temp<-cbind(as.factor(y),temp)
继续检查一下,以确保这个类是一个矩阵类型:
<class(temp)[1]"matrix"
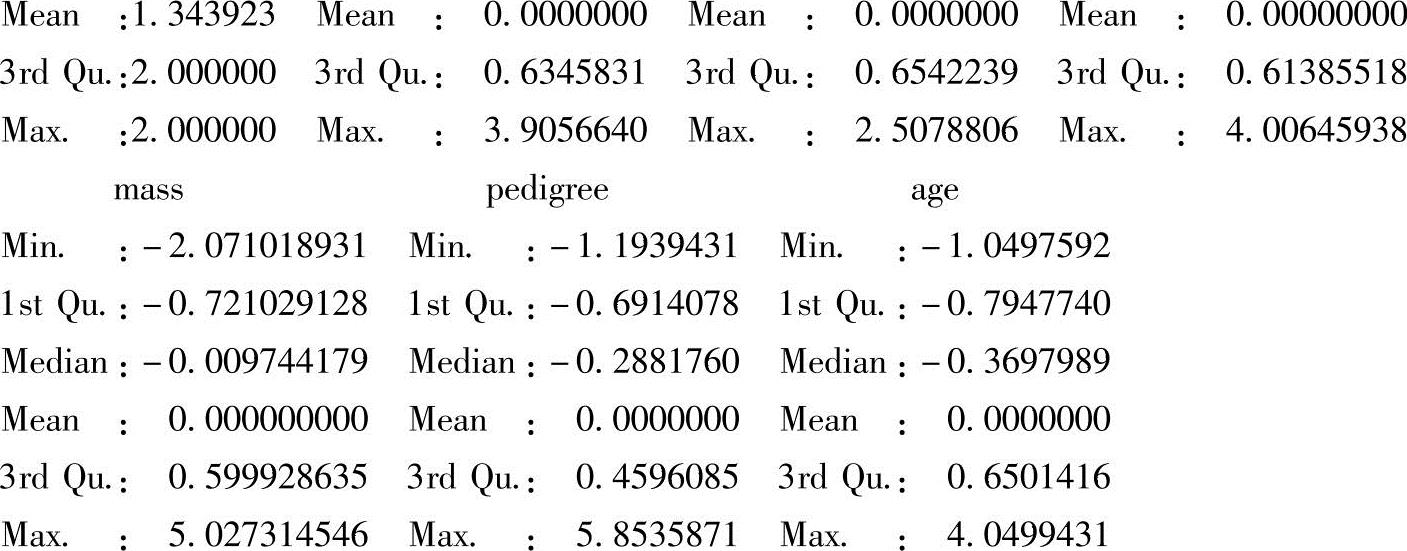
也可以使用summary,查看数据摘要:
>summary(temp)

最后,选择训练样本(724个观测值中的600个)。
<set.seed(2016)
<n=nrow(temp)(https://www.xing528.com)
<n_train<-600
<n_test<-n-n_train
<train<-sample(1:n,n_train,FALSE)
(2)使用RSNNS包建立DNN
<library(RSNNS)
为让问题尽可能简单,将响应变量赋值给Y,属性变量赋值给X。
<set.seed(2016)
<X<-temp[train,1:6]
<Y<-temp[train,7]
现在的问题是,如何在RSNNS包中指定分类DNN。
>fitMLP<-mlp(x=X,y=Y,
size=c(12,8),
maxit=1000,
initFunc="Randomize_Weights",
initFuncParams=c(-0.3,0.3),
learnFunc="Std_Backpropagation",
learnFuncParams=c(0.2,0),
updateFunc="Topological_Order",
updateFuncParams=c(0),
hiddenActFunc="Act_Logistic",
shufflePatterns=TRUE,
linOut=TRUE)
这个网络有两个隐藏层;第一个隐藏层包含12个神经元;第二隐含层包含8个神经元。使用隐藏层中的逻辑激活函数和输出层神经元的线性激活函数来随机地初始化权重和偏差。
注意到,对于这个数据集,R代码执行相对较快。
(3)使用predict函数进行预测
>predMLP<-sign(predict(fitMLP,temp[-train,1:6]))
既然有了分类数据,就应该查看其混淆矩阵。
<table(predMLP,sign(temp[-train,7]),dnn=c("Predicted","Observed"))
Observed
Predicted -1 1
-1 67 9
1 21 27
注意:模型不能完美地拟合数据。混淆矩阵中的对角线元素表示分类错误的数据数量。将错误分类的数据计算为错误率通常是有帮助的。做法如下:
<error_rate=(1-sum(predMLP==sign(temp[-train,7]))/124)
<round(error_rate,3)
[1]0.242
分类DNN具有大约24%的总体错误率(或大约76%的准确率)。
要强调的是,使用R可以轻松地、快速地构建分类DNN。如果使用C++和C编写代码要许多行,才可以完成现在R中几行代码就可以实现事情。如果读者对数据科学感兴趣,并且希望使用相关学科的工具来提取有意义的信息,那么使用R肯定会事半功倍。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。




