在英国,天气总是一个聊谈的话题,因为,英格兰的天气总是在变化。你可以在一天里体验四个季节。
【例4.1】使用Jordan网络建立诺丁汉市气温预测模型。
与Elman网络类似,Jordan网络模型也是对时间序列数据的建模工具。
(1)加载使用的包和数据
>library(RSNNS)
>library(quantmod)
>library(datasets)
>data(nottem)
数据是datasets包的nottem数据集,nottem数据集包含了诺丁汉市,每年每月平均测量空气温度。首先了解一下nottem的数据结构:
>nottem
没有出现任何缺失值,检查一下nottem数据类型:
>class(nottem)
[1]"ts"
Nottem是一个时间序列ts对象,了解数据的类型是非常重要的,特别是使用日期类型或R的不同类型的混合。
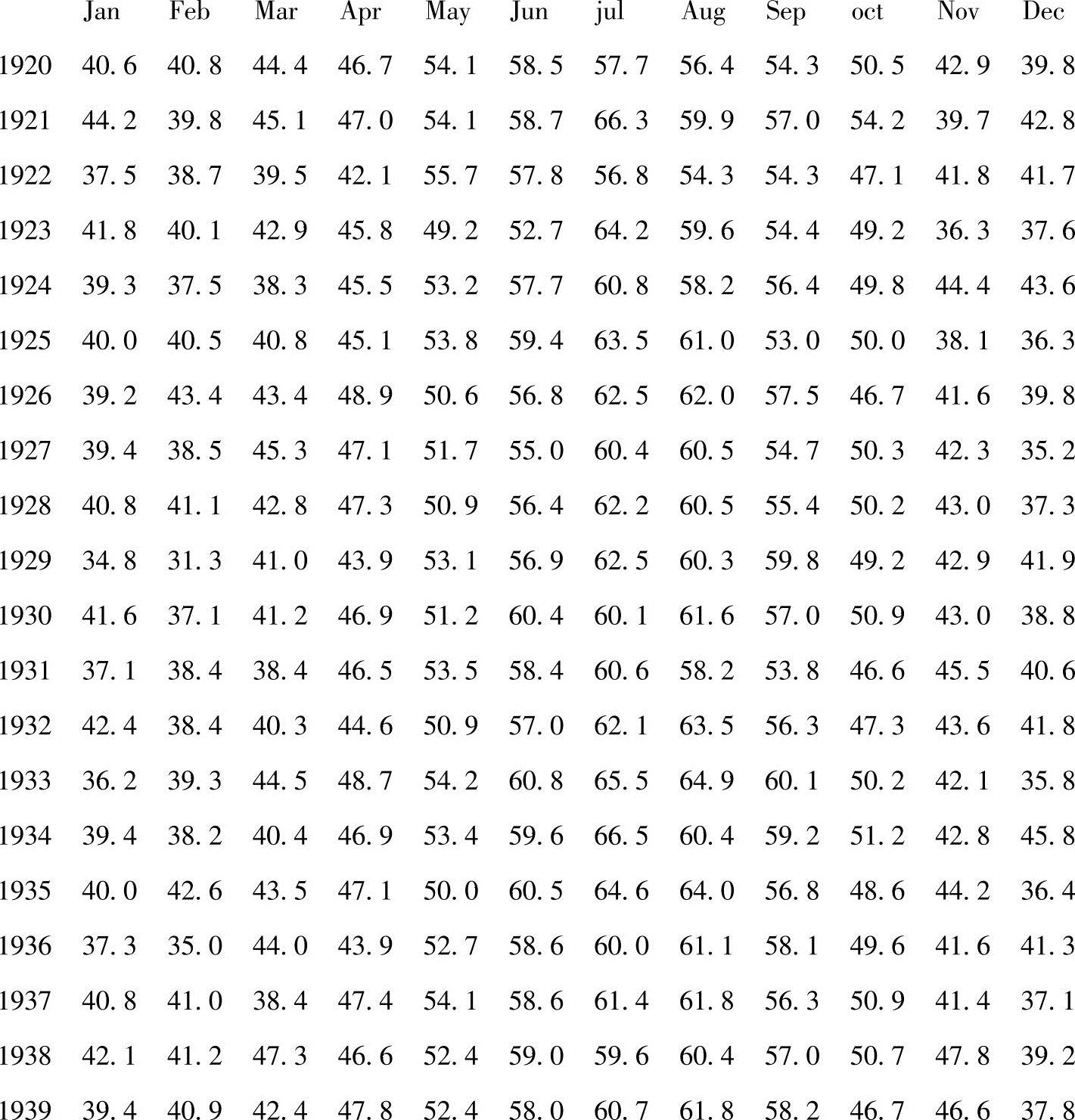
图4.6显示了nottem观察数据的时间序列散点图。数据覆盖1920~1939年,虽然没有任何明显趋势,然而它确实表现出强烈的季节性。
图4.6 1920~1939年诺丁汉市月平均温度
画时间序列散点图命令如下:
>plot(nottem)
(2)数据探索
在使用神经网络模型之前,需要归一化数据的属性。下面给出常用的四种归一化方法:
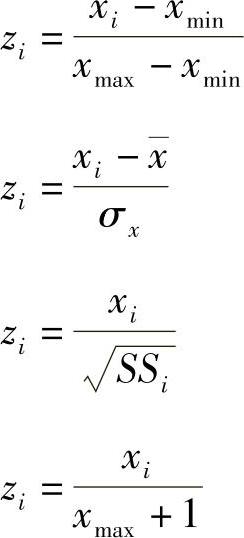
式中,SSi是xi的平方和;x-是xi的均值;σx是xi的标准差。因为没有任何说明性变量,所以,首先使用log变换数据,再使用scale函数标准化数据。
>y<-as.ts(nottem)
>y<-log(y)
>y<-as.ts(scale(y))
因为建模数据有很强的季节性特征,似乎依赖于月,所以使用滞后12个月的数据作为Jordan网络12个属性的输入。Quantmod包的Lag函数需要的数据类型为zoo类。
>y<-as.zoo(y)
>x1<-Lag(y,k=1)
>x2<-Lag(y,k=2)
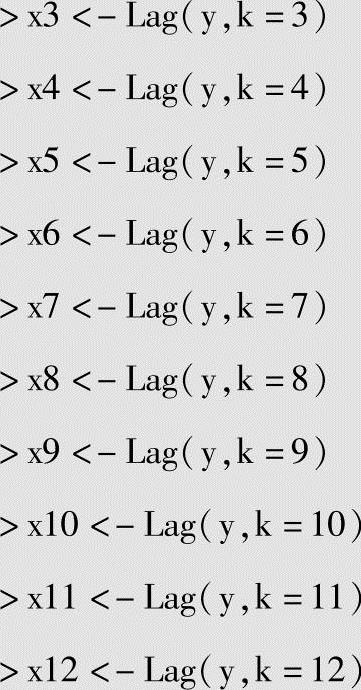
与Elman神经网络类似,需要删除NA值,最后将清洗后的数据存入temp中。
数据探索最后一步是使用plot函数可视化所有属性和响应变量的分布,如图4.7所示。
>plot(temp)
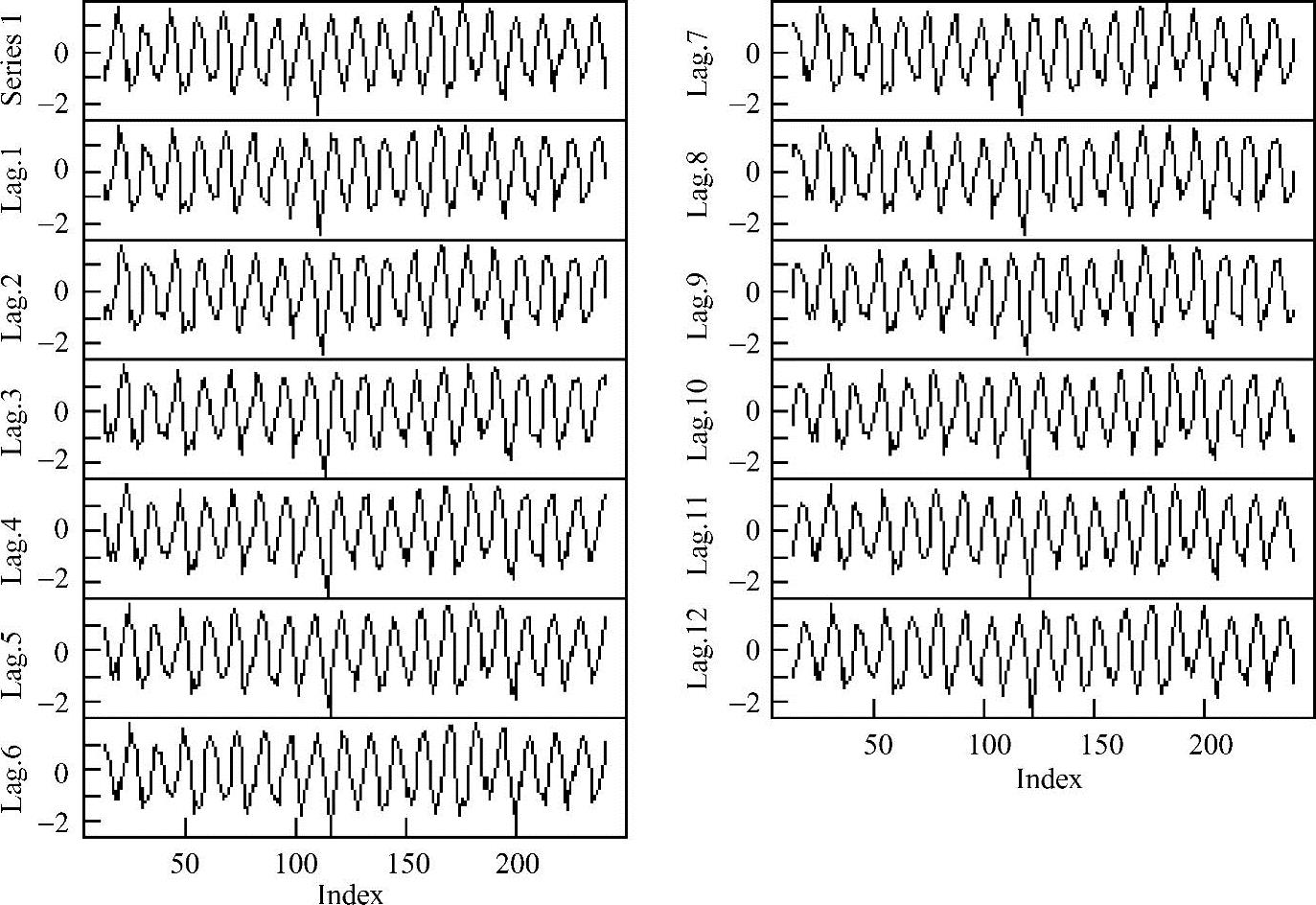
图4.7 Jordan网络响应变量和属性分布图
(3)训练样本选择
>n=nrow(temp) #观测数据个数
>n
[1]228
>set.seed(465) #设置种子
>n_train<-190#设置训练集大小
>train<-sample(1:n,n_train,FALSE) #随机选取190个训练样本
(4)建模
>inputs<-temp[,2:13] #属性变量
>outputs<-temp[,1] #响应变量
>fit<-jordan(inputs[train],outputs[train],#建模
size=2, #隐藏层数
learnFuncParams=c(0.01), #学习率
maxit=1000) #最大迭代次数
(5)模型部署
使用plotIterativeError函数显示迭代误差,如图4.8所示。
>plotIterativeError(fit)
前100次迭代误差下降相当快,大约迭代300次趋于稳定。
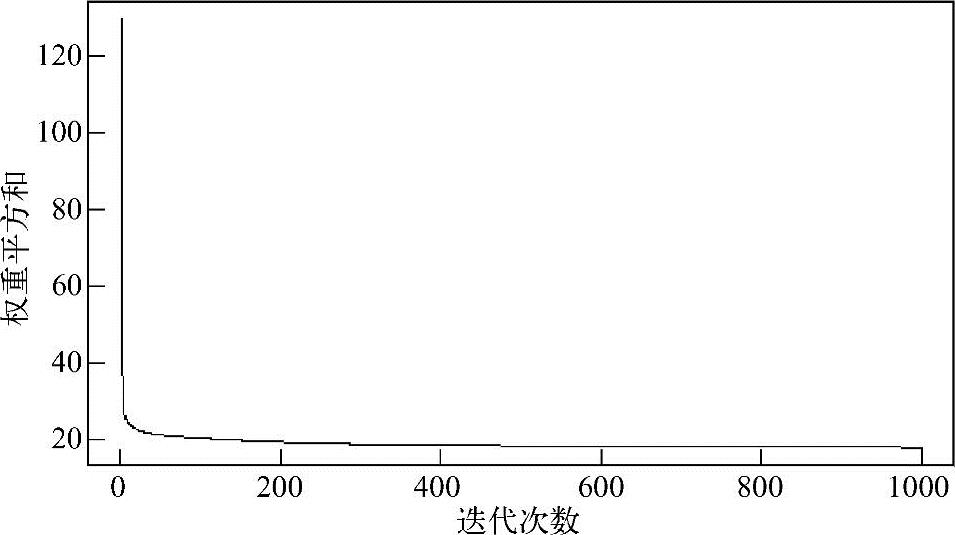
图4.8 训练误差与迭代次数
使用predict函数对测试样本进行预测,并计算测试样本响应变量和预测值之间的平方相关系数。
>pred<-predict(fit,inputs[-train])
>cor(outputs[-train],pred)^2
[1,]0.9050079
因为平方相关系数大于0.9,所以模型是成功的。
【例4.2】建立一个RNN模型,预测在英国死于支气管炎、肺气肿、哮喘的总人数。
(1)检查机器上安装了哪些包
>pack<-as.data.frame(installed.packages()[,c(1,3:4)])
>rownames(pack)<-NULL
>pack<-pack[is.na(pack$Priority),1:2,drop=FALSE]
>print(pack,row.names=FALSE)
(2)加载需要的包和数据
>library(RSNNS)
>library(quantmod)
>library(datasets)
>data(UKLungDeaths)
使用的数据在datasets包里面,所需要的数据结构为UKLungDeaths。dataests包是默认加载的包,所以不需要专门加载它。但是,调用它也是一种有益的练习。
UKLungDeaths数据库包含了3个1974~1979年英国每个月支气管炎、肺气肿、哮喘的死亡人数的时间序列。第一个时间序列是死亡的总人数(ldeaths);第二个时间序列是男性死亡人数(mdeaths);第三个时间序列是女性死亡人数(fmdeaths)。可以通过使用plot函数可视化这些数据(见图4.9),方法如下:
>par(mfrow=c(1,3)) #窗口划分为1行3列
>plot(ldeaths,xlab="Year",ylab="Both sexes",main="Total")
>plot(mdeaths,xlab="Year",ylab="Males",main="Males")
>plot(fdeaths,xlab="Year",ylab="Females",main="Females")
总而言之,随着时间的推移,可以看到死亡人数有着巨大的变化,每月死亡人数的波动清晰可见。可以观察到,1976年出现每月最高和最低的死亡总数。有趣的是,对于男性来说,每个周期的最低点呈现下降的趋势。而进入1979年以后所有的趋势都呈相当明显的下降。
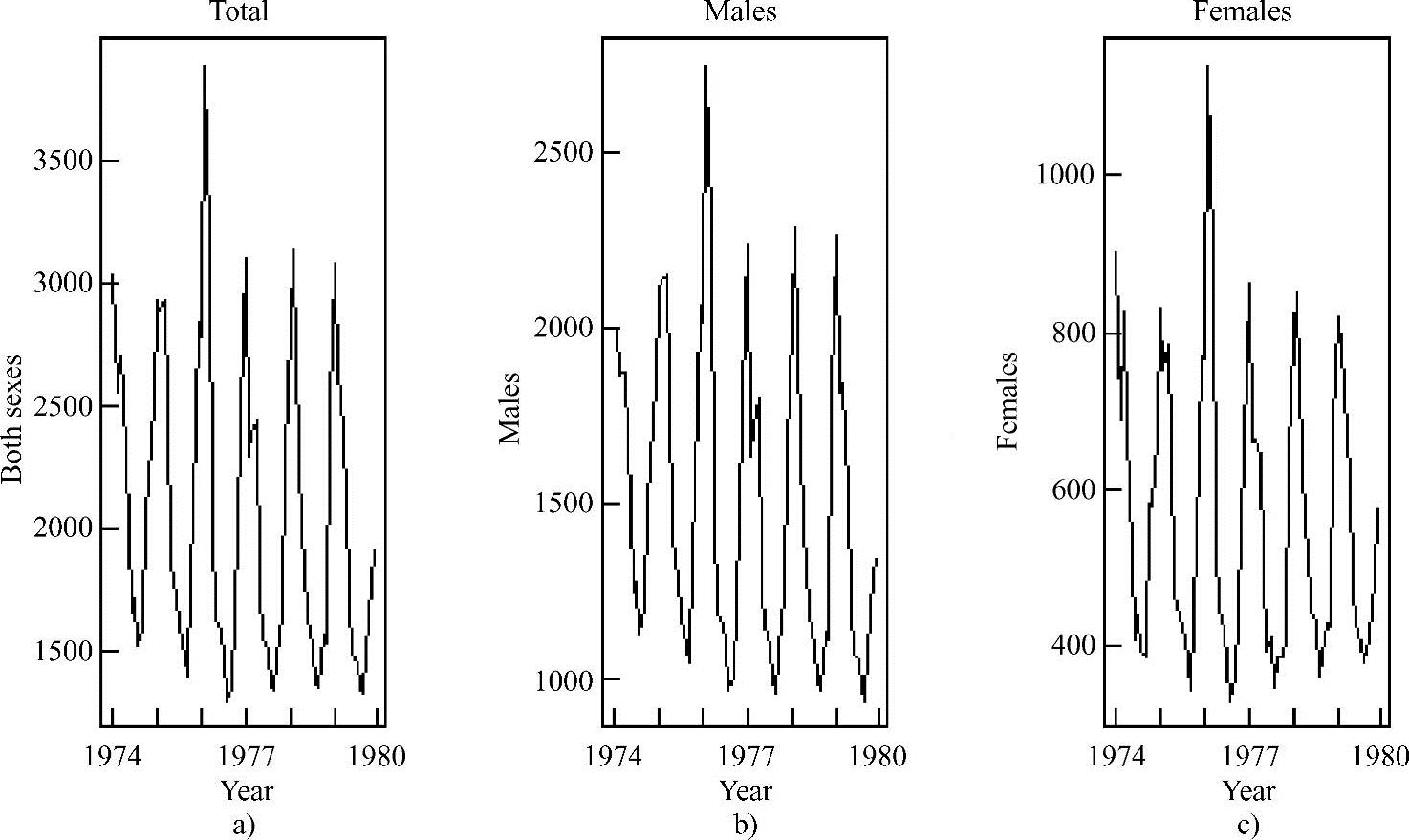
图4.9 英国每个月支气管炎、肺气肿、哮喘死亡人数
a)总人数 b)男性死亡人数;c)女性死亡人数(https://www.xing528.com)
(3)数据探索
因为对死亡总数的建模感兴趣,所以重点分析ldeaths数据结构。在此之前,快速检查缺失数据。
>sum(is.na(ldeaths))
[1]0
表示数据集ldeaths没有缺失数据,接下来,检查ldeaths类型。
>class(ldeaths)
[1]"ts"
表示ldeaths是一个时间序列对象,图4.10显示了时间序列图、核密度图和箱线图。
>par(mfrow=c(3,1)) #窗口划分为3行1列
>plot(ldeaths)
>x<-density(ldeaths)
>plot(x,main="UK total deaths from lung diseases")
>polygon(x,col="green",border="black")
>boxplot(ldeaths,col="cyan",ylab="NumbeR of deaths per month")
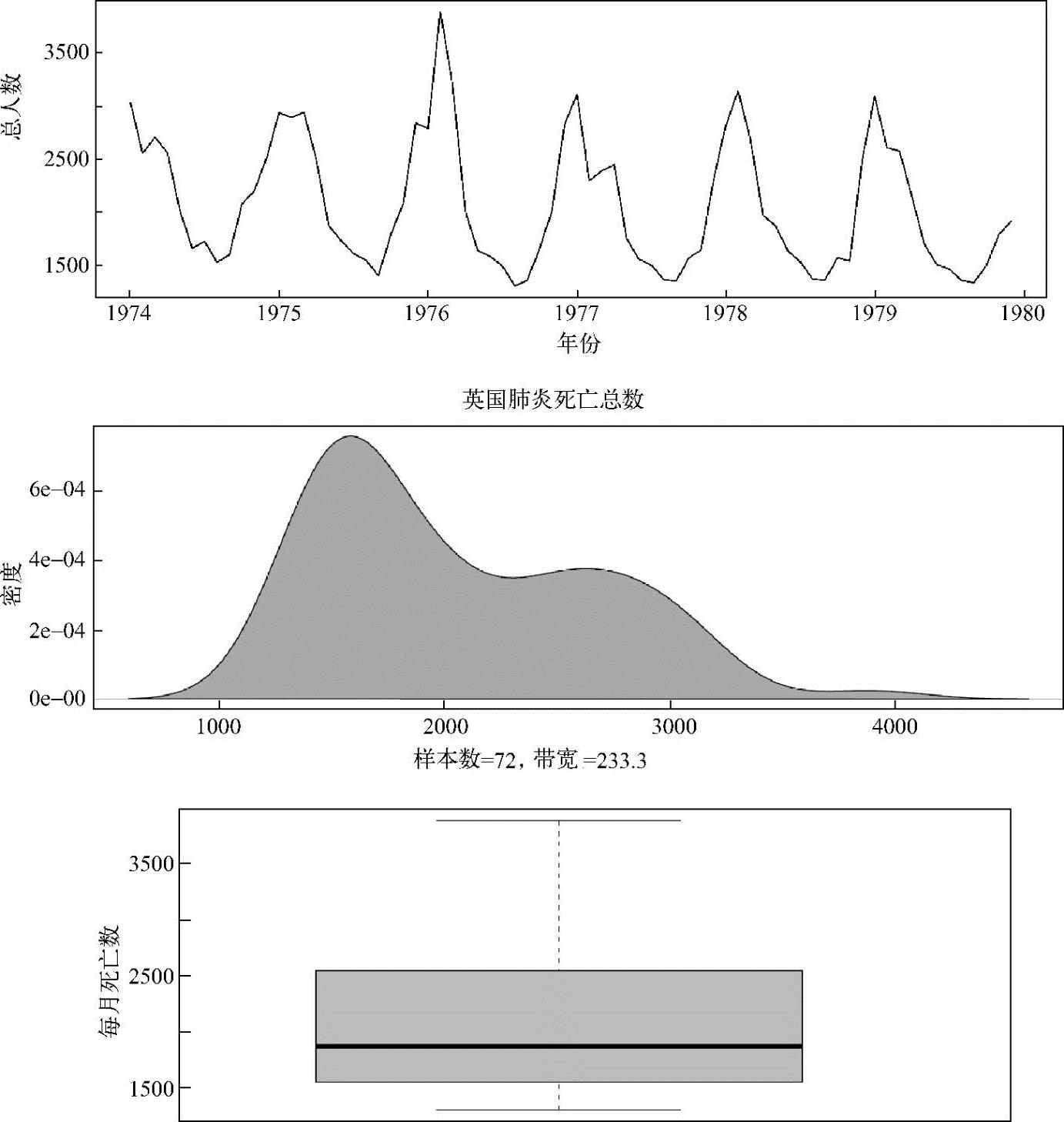
图4.10 死亡总数的形象化总结
(4)数据转换
由于数据似乎存在某种规律,所以将其转换成一种适合进行Elman神经网络工作的形式。首先,将数据复制一份,存储在变量y中。
>y<-as.ts(ldeaths)
当使用时间序列时,一般将数据转换成对数。将数据转换成对数,数据会更加规范化。
>y<-log(y)
通过scale函数使数据看起来更加规范。
>y<-as.ts(scale(y))
注意,as.ts函数确保变换后仍然是ts对象。
既然无法将这个数据归于其他的类别,那么就使用纯时间序列的方法。这种建模方法的主要问题是需要多少滞后变量(lags)?既然是对一年内的每个月都进行了观察,所以使用12作为滞后变量。实现这个操作的方法是使用quantmod包里面的Lag操作函数,这就需要将y转变成一个zoo类的对象。
>y<-as.zoo(y)
x1<-Lag(y,k=1)
x2<-Lag(y,k=2)
x3<-Lag(y,k=3)
x4<-Lag(y,k=4)
x5<-Lag(y,k=5)
x6<-Lag(y,k=6)
x7<-Lag(y,k=7)
x8<-Lag(y,k=8)
x9<-Lag(y,k=9)
x10<-Lag(y,k=10)
x11<-Lag(y,k=11)
x12<-Lag(y,k=12)
通过这个操作得到了12个属性作为神经网络的输入。下一步是将这12个观测值组合成一个数据框。
>deaths<-cbind(x1,x2,x3,x4,x5,x6,x7,x8,x9,x10,x11,x12)>deaths<-cbind(y,deaths)观察deaths里面包含什么。
>head(round(deaths,2),10)
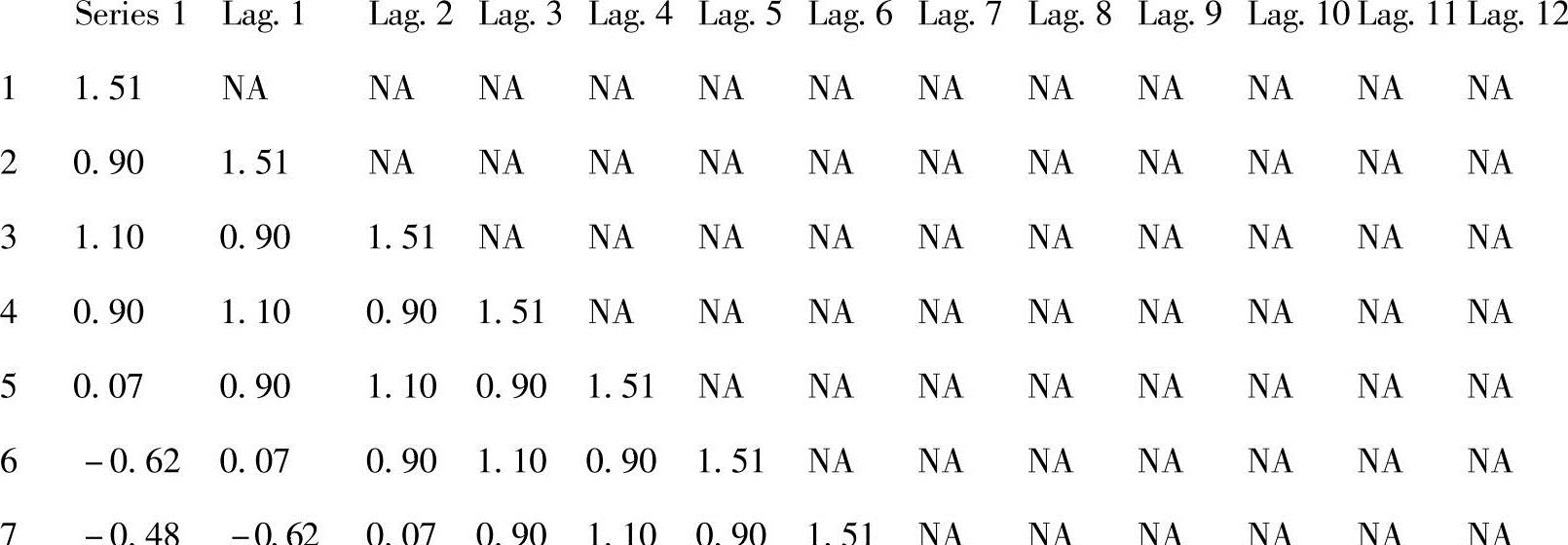
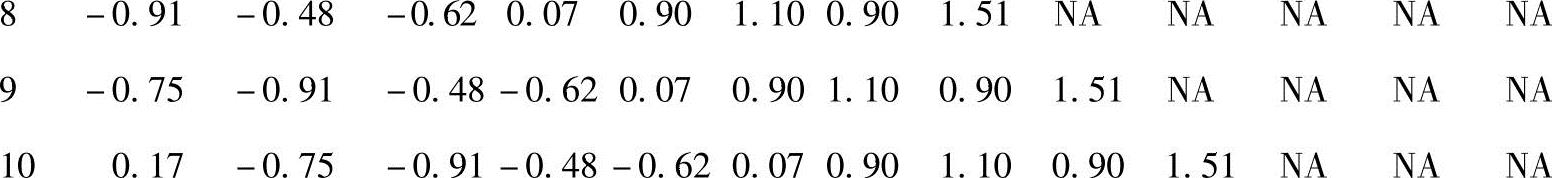
注意到NA的滞后值从1增大到12。使用延迟的目的就是为了得到这个结果。然而,需要从数据集里面删去NA观测值。
>deaths<-deaths[-(1:12),]
现在已经准备好开始创建训练集和测试集。首先,计算出数据的行数,并且使用set.seed函数来输出它。
>n=nrow(deaths)
>n
[1]60
>set.seed(465)
作为一种快速的检测方法,可以看到有60行的观测数据可以用来作为分析。为了解决滞后值的问题,删除了12行的观测值,而结果也达到了预期。
使用其中的45行数据来建模,剩下的15行用来作为测试集。用如下代码随机选择一些不重复的数据:
>n_train<-45
>train<-sample(1:n,n_train,FALSE)
(5)建模
为了使过程变得更加简单一点,可以将包含有滞后值的协方差的属性放入到R的inputs对象里面,将相应变量放到ouputs对象里面。
>inputs<-deaths[,2:13]
>outputs<-deaths[,1]
在每个神经网络中,配置有两个隐藏层,每个隐藏层里面都包含一个结点。并将学习速率设置为0.1,最大的迭代次数设置为1000。
>fit<-elman(inputs[train],outputs[train],size=c(1,1),
leaRnFuncPaRams=c(0.1),maxit=1000)
若给定一个较小的数据,模型的收敛会变得很快,可以画出如图4.11所示的误差函数。
>plotIterativeError(fit)
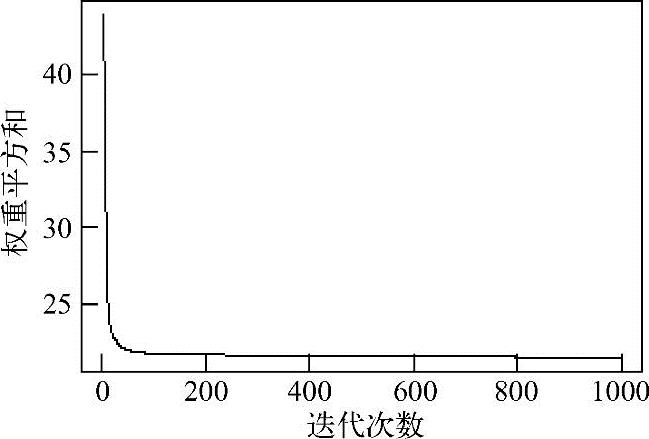
图4.11 Elman函数误差图像
从图4.11可以看出,误差迅速减小,大约在500次迭代后趋于稳定,可以使用summaRy函数来得到神经网络细节方面的信息。
>summary(fit)通过这个函数可以得到很多R的输出值,找到输出中如下所示的部分。
unit definition seclion:
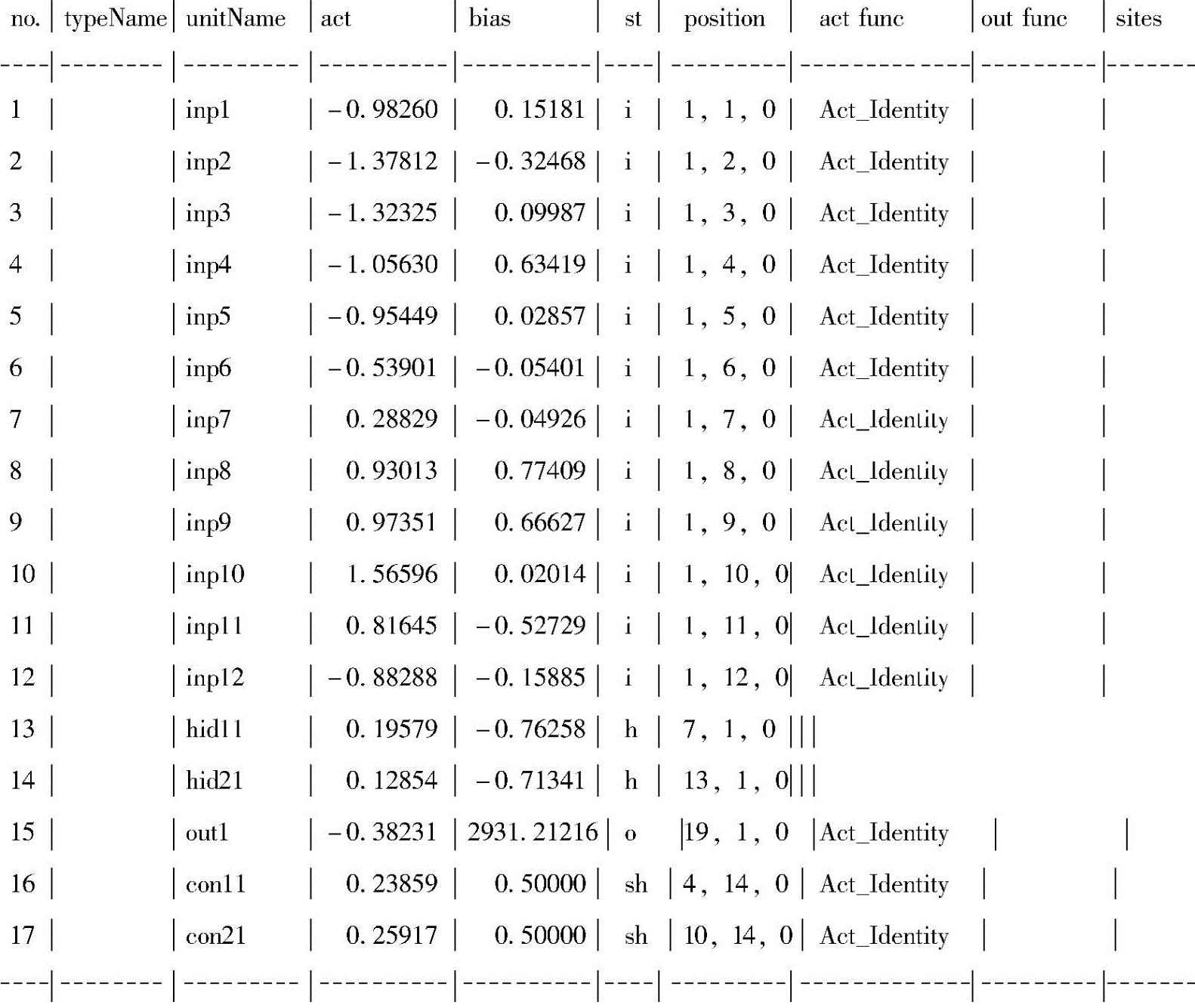
从第一列中,可以得到结点或者神经元的数量,而且可以知道这个网络一共有17行。它的第三列描述了神经元的类型,从中可以得知有12个输入神经元、2个隐藏神经元和1个输出神经元,在承接层有2个神经元。第四列和第五列给出了激活函数的值和每个神经元的偏置。例如,第一个神经元的激活函数值为-0.98260,偏置为0.15181。
(6)模型部署
现在,在测试集上用prRedict函数来测试一下这个模型。
>pred<-predict(fit,inputs[-train])
>plot(outputs[-train],pred)
(7)模型评估
预测值和实际值的散点图如图4.12所示。
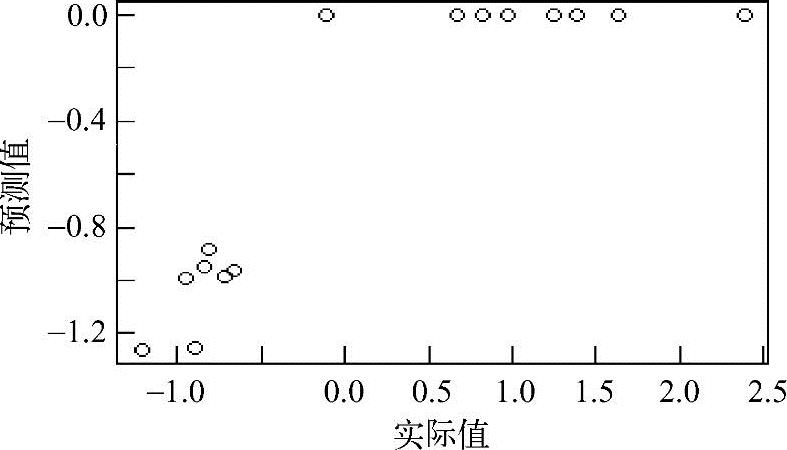
图4.12 RNN的实际值和预测值
>cor(outputs[-train],pred)^2
[,1]
[1,]0.7845
由此可知,平方相关系数为0.78。可以尝试不同的模型来提高这个系数,以达到理想值。试一试,通过重新建模来达到整体性能提高的效果。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。




